研究組織・メンバー
A02:生体脳内ゲノム編集によるハイスループットな細胞種識別技術の開発と応用
 研究代表者
研究代表者
三國 貴康
新潟大学脳研究所・教授
WEBhttps://www.bri.niigata-u.ac.jp/~cellular_neuropath/
紹介文本文
哺乳類の脳は、様々な細胞種同士が互いに複雑なネットワークを形成している、極めてヘテロで難解なシステムである。従って、脳の動作原理を抽出するためには、脳の中の特定の細胞種を識別し、細胞種選択的に神経活動をモニターする必要がある。
細胞種を識別するための遺伝子組換えマウスの系統の作製には、時間とコストがかかる。また、複数の細胞種を識別するには異なる系統のマウスの交配が必要であり、さらに、病態モデルマウスに適用するにはより多くの交配が必要になるので、現実的には困難である。加えて、マウス以外の哺乳類での動物系統の作製は、未だに技術的に困難な点が多い。
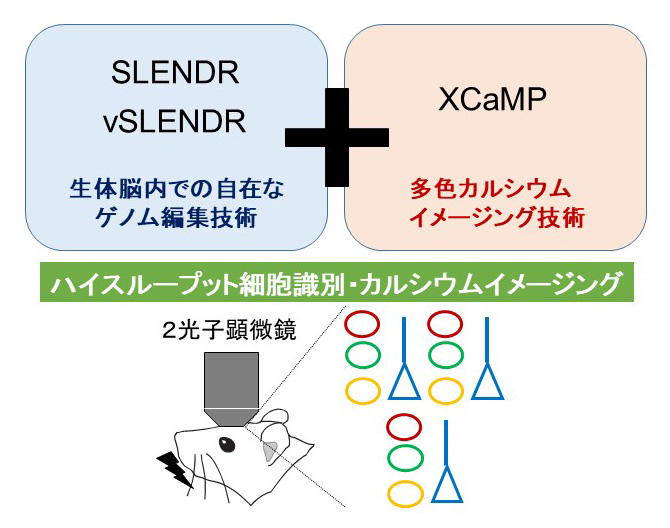
三國はこれまでに、生体脳内で迅速かつ正確なゲノム編集を可能にする技術(SLENDR法とvSLENDR法)を開発してきた (Mikuni et al., Cell 2016; Nishiyama*, Mikuni* et al., Neuron 2017)。SLENDR/vSLENDR法により、あらゆる時期の個体の脳の任意の細胞種、脳領域あるいは脳全体で効率良く自在にゲノムを編集できる。
そこで、本研究では、SLENDR/vSLENDR法を駆使して、動物個体の脳でハイスループットに細胞種を識別し、識別した細胞種の神経活動をイメージングするための技術基盤を開発する。当領域で開発された世界最高感度の多色のカルシウムインジケーターXCaMP技術と組み合わせることで、ハイスループットに複数の細胞種で神経活動を同時にモニターする。これにより、ヘテロで難解なシステムである脳の動作原理の解明につなげることを目指す。
文献
- Mikuni T, Uchigashima M (2020)
Methodological approaches to understand the molecular mechanism for structural plasticity of dendritic spines.
Eur J Neurosci: in press. - Nishiyama J, Mikuni T, Yasuda R (2017)
Virus-mediated genome editing via homology-directed repair in mitotic and postmitotic cells in mammalian brain.
Neuron 96(4): 755-68. - Mikuni T, Nishiyama J, Sun Y, Kamasawa N, Yasuda R (2016)
High-throughput, high-resolution mapping of protein localization in mammalian brain by in vivo genome editing.
Cell 165(7): 1803-17. - Mikuni T, Uesaka N, Okuno H, Hirai H, Deisseroth K, Bito H, Kano M (2013)
Arc/Arg3.1 is a postsynaptic mediator of activity-dependent synapse elimination in the developing cerebellum.
Neuron 78(6): 1024-35.


